The Lattes platform has been
hosting curricula of Brazilian researchers since the late 1990s,
containing more than 5 million curricula. The data from the Lattes
curricula can be downloaded to XML format, the complexity
of this reading process motivated the development of the
getLattes package, which imports the information from the
XML files to a list in the R software and then
tabulates the Lattes data to a data.frame.
The main information contained in XML files, and
imported via getLattes, are:
- Research Area
getAreasAtuacao()
- Published Papers
getArtigosPublicados()
- Accepted Papers
getArtigosAceitos()
- Profissional Links
getAtuacoesProfissionais()
- Ph.D. Examination Board’s
getBancasDoutorado()
- Undergraduate Examination Board’s
getBancasGraduacao()
- Master Examination Board’s
getBancasMestrado()
- Books Chapters
getCapitulosLivros()
- General Data
getDadosGerais()
- Profissional Address
getEnderecoProfissional()
- Events and Congresses
getEventosCongressos()
- Profissional Formation (Ph.D. Thesis)
getFormacaoDoutorado()
- Profissional Formation (Master Thesis)
getFormacaoMestrado()
- Profissional Formation (Undergraduation)
getFormacaoGraduacao()
- Languages
getIdiomas()
- Research Lines
getLinhaPesquisa()
- Published Books
getLivrosPublicados()
- Event’s Organization
getOrganizacaoEvento() - Academic Advisory (Ph.D. Thesis)
getOrientacoesDoutorado()
- Academic Advisory (Master Thesis)
getOrientacoesMestrado()
- Academic Advisory (Post Doctorate)
getOrientacoesPosDoutorado()
- Other Technical Productions
getOutrasProducoesTecnicas()
- Participation in Projects
getParticipacaoProjeto()
- Technical Production
getProducaoTecnica()
- Patents
getPatentes()
- Patents
getTrabalhosEmEventos()
- Personal Lattes 16 digits identification
getId()
From the functionalities presented in this package, the main
challenge to work with the Lattes curriculum data is now to download the
data, as there are Captchas. To download a lot of curricula I suggest
the use of Captchas Negated
by Python reQuests - CNPQ. The second barrier to be overcome is the
management and processing of a large volume of data, the whole Lattes
platform in XML files totals over 200 GB. In this tutorial
we will focus on the getLattes package features, being the
reader responsible for download and manage the files.
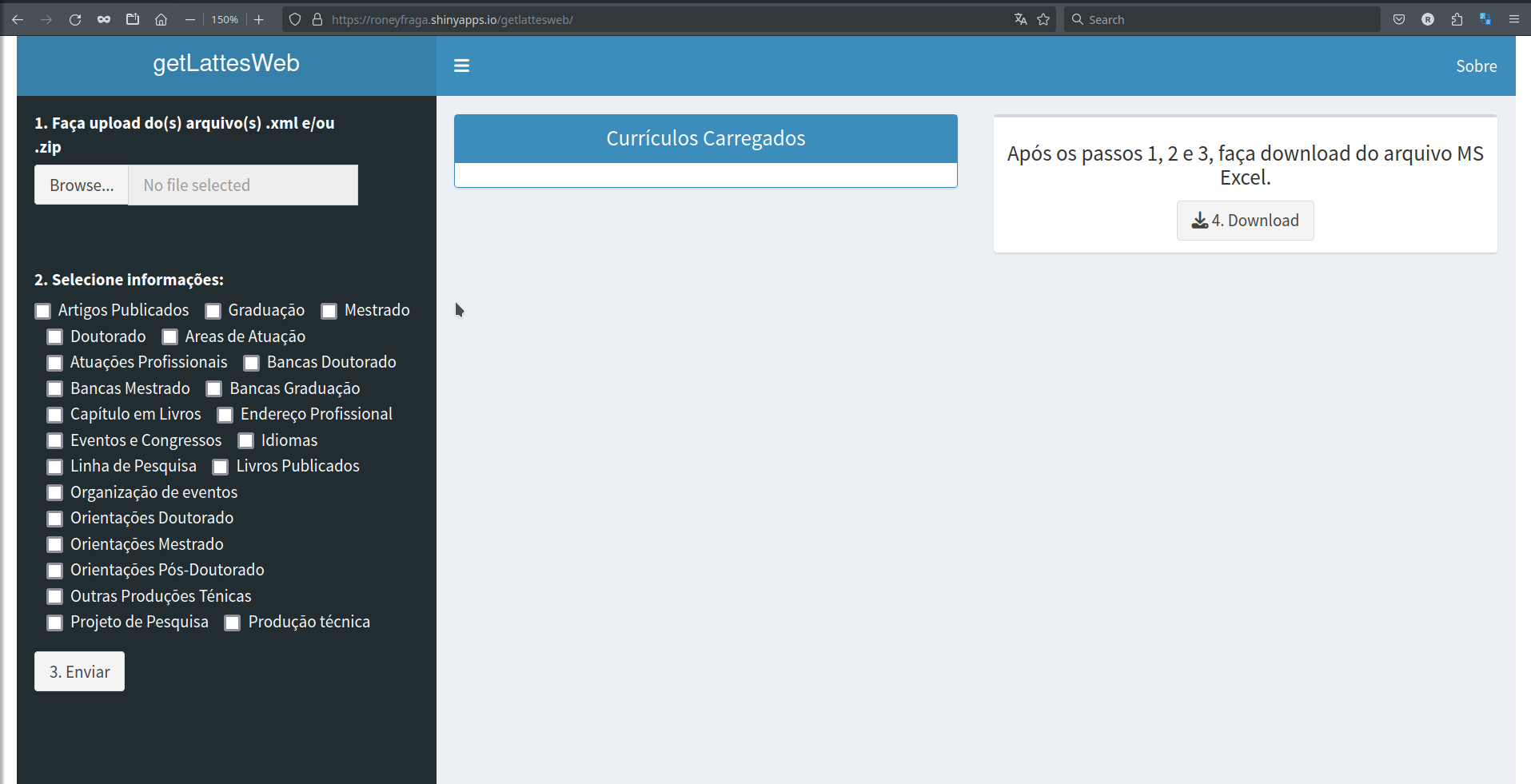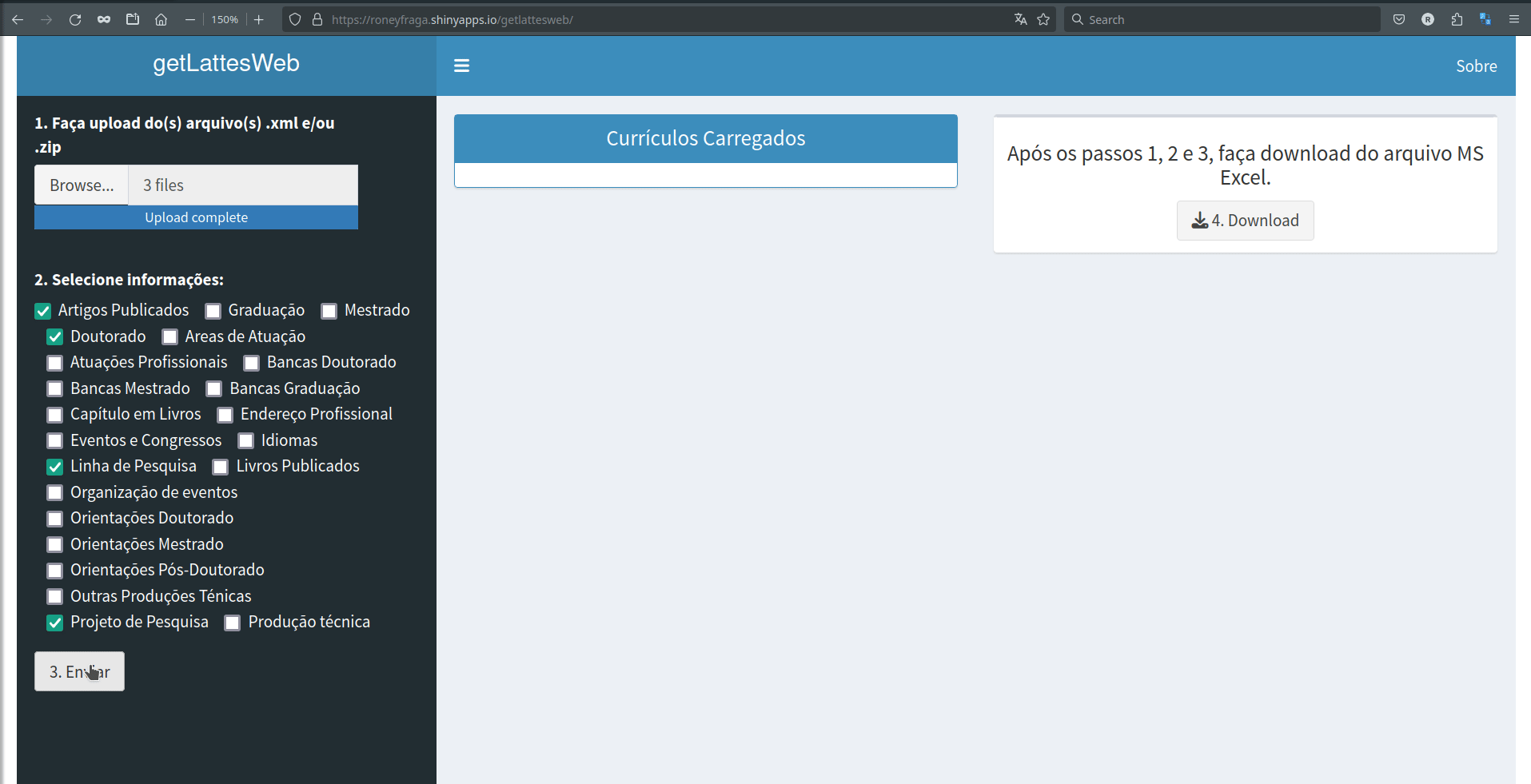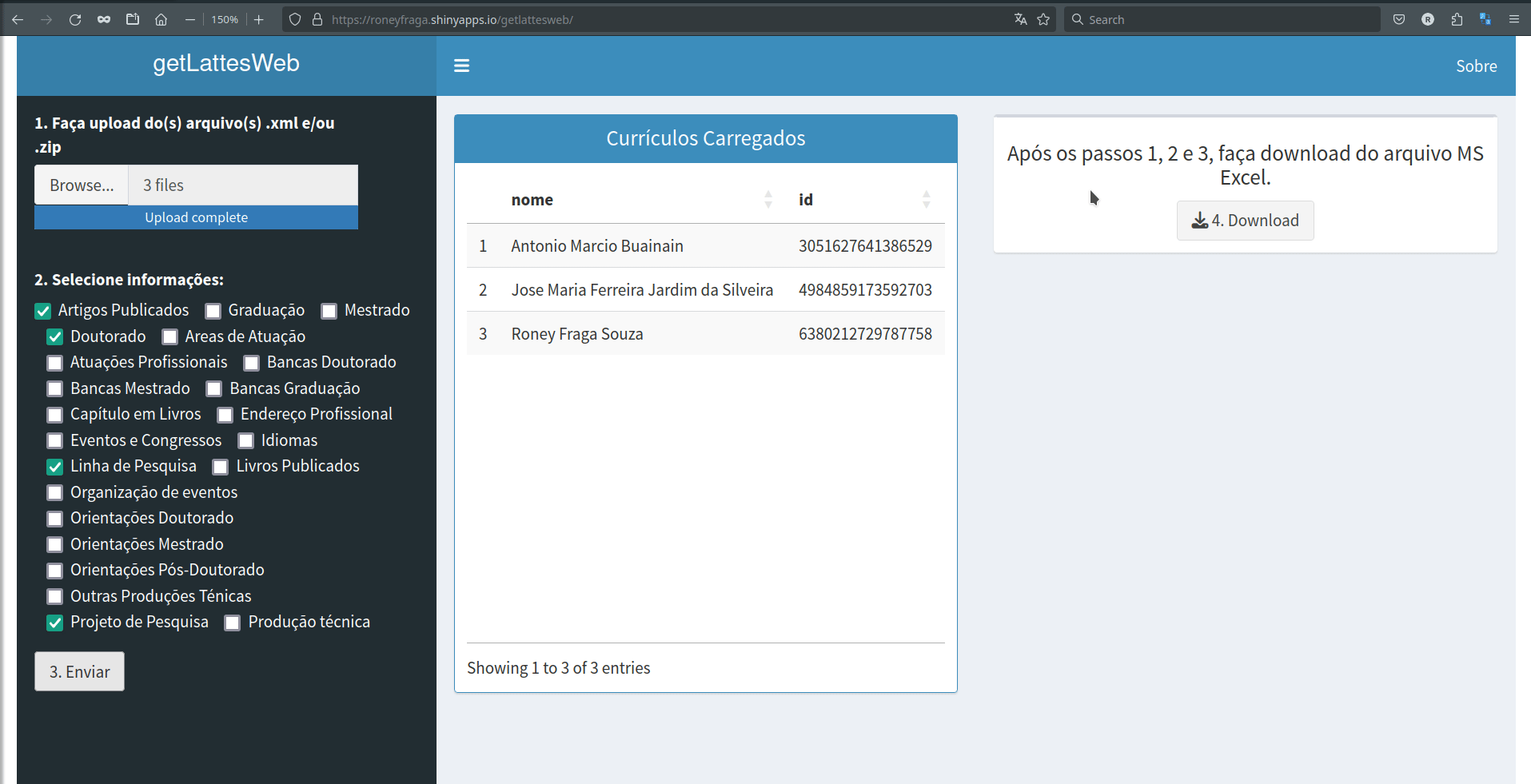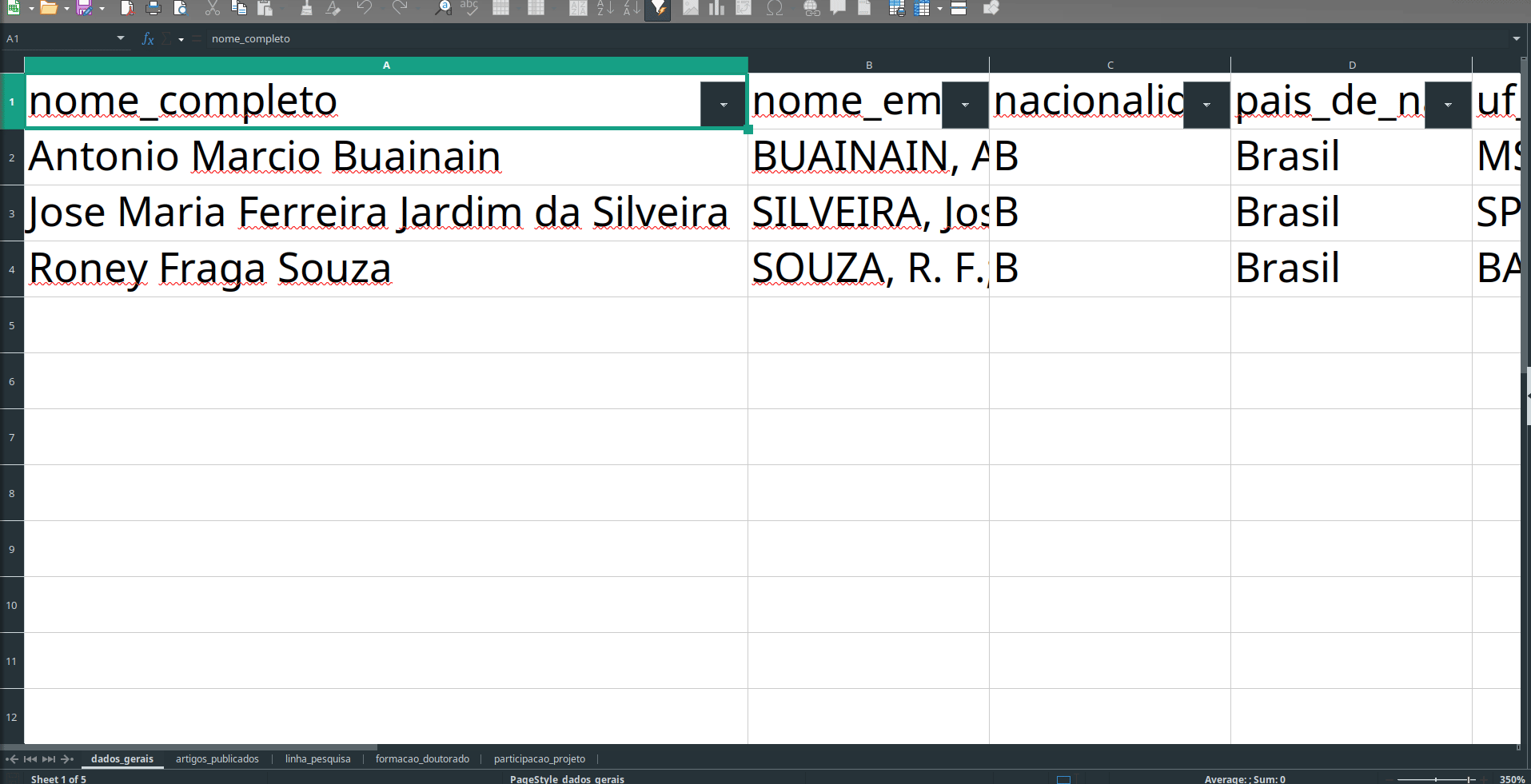
Follow an example of how to search and download data from the Lattes website.
getLattesWeb
Alternative for no-coders:
- link 1 https://roneyfraga.shinyapps.io/getlattesweb/
- link 2 http://roneyfraga.com/shiny/getLattesWeb/
Installation
To install the newest released version of getLattes from github.
# install and load devtools from CRAN
# install.packages("devtools")
library(devtools)
# install and load getLattes
devtools::install_github("roneyfraga/getLattes")Stable version from CRAN.
install.packages('getLattes')Load getLattes.
Single curriculum
Import
Using the get* functions to import data from a single
curriculum is straightforward. The curriculum need to be imported into
R by the read_xml() function from the
xml2 package.
# find the file in system
zip_xml <- system.file('extdata/4984859173592703.zip', package = 'getLattes')
curriculo <- xml2::read_xml(zip_xml)
get functions
getDadosGerais(curriculo)
getAreasAtuacao(curriculo)
getArtigosPublicados(curriculo)
getAtuacoesProfissionais(curriculo)
getBancasDoutorado(curriculo)
getBancasGraduacao(curriculo)
getBancasMestrado(curriculo)
getCapitulosLivros(curriculo)
getDadosGerais(curriculo)
getEnderecoProfissional(curriculo)
getEventosCongressos(curriculo)
getFormacaoDoutorado(curriculo)
getFormacaoGraduacao(curriculo)
getFormacaoMestrado(curriculo)
getIdiomas(curriculo)
getLinhaPesquisa(curriculo)
getLivrosPublicados(curriculo)
getOrganizacaoEventos(curriculo)
getOrientacoesDoutorado(curriculo)
getOrientacoesMestrado(curriculo)
getOrientacoesPosDoutorado(curriculo)
getOutrasProducoesTecnicas(curriculo)
getParticipacaoProjeto(curriculo)
getPatentes()
getProducaoTecnica(curriculo)
getTrabalhosEmEventos()
getId(curriculo)Several curricula
Import
To import data from two or more curricula it is easier to use
list.files(), a native R function, or dir_ls()
from fs package. As xml2::read_xml() allow to
read a xml curriculum inside a zip files.
# find the files in system
zips_xmls <- c(system.file('extdata/4984859173592703.zip', package = 'getLattes'),
system.file('extdata/3051627641386529.zip', package = 'getLattes'))Import the listed curricula to R memory as
xml2::read_xml object.
curriculos <- lapply(zips_xmls, read_xml)The lapply() function is a well-known and widely used
alternative in the R world. However, it does not natively
handle errors, which makes the map function from the
purrr package an excellent alternative.
Adding an extra layer of complexity, I will use pipe
|>. Programming using the pipe operator
|> allows faster coding and clearer syntax.
get functions
To read data from only one curriculum any function get
can be executed singly, but to import data from two or more curricula is
easier to use get* functions with lapply() or
map().
dados_gerais <-
purrr::map(curriculos, safely(getDadosGerais)) |>
purrr::map(pluck, 'result')
dados_gerais
#> [[1]]
#> # A tibble: 1 × 12
#> nome_completo nome_em_citacoes_bib…¹ nacionalidade pais_de_nascimento
#> <chr> <chr> <chr> <chr>
#> 1 Jose Maria Ferreira J… SILVEIRA, José Maria … B Brasil
#> # ℹ abbreviated name: ¹nome_em_citacoes_bibliograficas
#> # ℹ 8 more variables: uf_nascimento <chr>, cidade_nascimento <chr>,
#> # permissao_de_divulgacao <chr>, data_falecimento <chr>,
#> # sigla_pais_nacionalidade <chr>, pais_de_nacionalidade <chr>,
#> # orcid_id <chr>, id <chr>
#>
#> [[2]]
#> # A tibble: 1 × 12
#> nome_completo nome_em_citacoes_bib…¹ nacionalidade pais_de_nascimento
#> <chr> <chr> <chr> <chr>
#> 1 Antonio Marcio Buaina… BUAINAIN, Antonio Mar… B Brasil
#> # ℹ abbreviated name: ¹nome_em_citacoes_bibliograficas
#> # ℹ 8 more variables: uf_nascimento <chr>, cidade_nascimento <chr>,
#> # permissao_de_divulgacao <chr>, data_falecimento <chr>,
#> # sigla_pais_nacionalidade <chr>, pais_de_nacionalidade <chr>,
#> # orcid_id <chr>, id <chr>Import general data from 2 curricula. The output is a list of data
frames, converted by a unique data frame with
bind_rows().
dados_gerais <-
purrr::map(curriculos, safely(getDadosGerais)) |>
purrr::map(pluck, 'result') |>
dplyr::bind_rows()
glimpse(dados_gerais)
#> Rows: 2
#> Columns: 12
#> $ nome_completo <chr> "Jose Maria Ferreira Jardim da Silveir…
#> $ nome_em_citacoes_bibliograficas <chr> "SILVEIRA, José Maria F. J.;Silveira, …
#> $ nacionalidade <chr> "B", "B"
#> $ pais_de_nascimento <chr> "Brasil", "Brasil"
#> $ uf_nascimento <chr> "SP", "MS"
#> $ cidade_nascimento <chr> "São Paulo", "Campo Grande"
#> $ permissao_de_divulgacao <chr> "NAO", "NAO"
#> $ data_falecimento <chr> "", ""
#> $ sigla_pais_nacionalidade <chr> "BRA", "BRA"
#> $ pais_de_nacionalidade <chr> "Brasil", "Brasil"
#> $ orcid_id <chr> "https://orcid.org/0000-0003-3680-875X…
#> $ id <chr> "4984859173592703", "3051627641386529"It is worth remembering that all variable names obtained by
get* functions are the transcription of the field names in
the XML file, the - being replaced with
_ and the capital letters replaced with lower case
letters.
Publications
artigos_publicados <-
purrr::map(curriculos, safely(getArtigosPublicados)) |>
purrr::map(pluck, 'result') |>
dplyr::bind_rows()
artigos_publicados |>
dplyr::arrange(desc(ano_do_artigo)) |>
dplyr::select(titulo_do_artigo, ano_do_artigo, titulo_do_periodico_ou_revista)
#> # A tibble: 192 × 3
#> titulo_do_artigo ano_do_artigo titulo_do_periodico_…¹
#> <chr> <chr> <chr>
#> 1 An Analysis of Collaboration Networks i… 2021 REVISTA DE ADMINISTRA…
#> 2 Patent network analysis in agriculture:… 2021 ECONOMICS OF INNOVATI…
#> 3 GENETICALLY MODIFIED CORN ADOPTION IN B… 2021 REVISTA DE ECONOMIA E…
#> 4 International trade in GMOs: have marke… 2020 Revista de economia e…
#> 5 Governance and financial efficiency of … 2020 RAUSP Management Jour…
#> 6 The Role of Participation in the Respon… 2020 Sustainability
#> 7 The impact of sugarcane expansion in Br… 2020 JOURNAL OF RURAL STUD…
#> 8 Innovation in GMOs, technological gap, … 2020 Agribusiness
#> 9 Avaliação do Programa Nacional de Produ… 2020 Desenvolvimento em De…
#> 10 Agro brasileiro em evolução: complexida… 2020 Revista de Política A…
#> # ℹ 182 more rows
#> # ℹ abbreviated name: ¹titulo_do_periodico_ou_revista
livros_publicados <-
purrr::map(curriculos, safely(getLivrosPublicados)) |>
purrr::map(pluck, 'result') |>
dplyr::bind_rows()
capitulos_livros <-
purrr::map(curriculos, safely(getCapitulosLivros)) |>
purrr::map(pluck, 'result') |>
dplyr::bind_rows() Grouping data
To group the data key variable is id, which is a unique
16 digit code.
artigos_publicados2 <-
dplyr::group_by(artigos_publicados, id) |>
dplyr::tally(name = 'artigos')
artigos_publicados2
#> # A tibble: 2 × 2
#> id artigos
#> <chr> <int>
#> 1 3051627641386529 101
#> 2 4984859173592703 91
livros_publicados2 <-
dplyr::group_by(livros_publicados, id) |>
dplyr::tally(name = 'livros')
livros_publicados2
#> # A tibble: 2 × 2
#> id livros
#> <chr> <int>
#> 1 3051627641386529 45
#> 2 4984859173592703 8
capitulos_livros2 <-
dplyr::group_by(capitulos_livros, id) |>
dplyr::tally(name = 'capitulos')
capitulos_livros2
#> # A tibble: 2 × 2
#> id capitulos
#> <chr> <int>
#> 1 3051627641386529 81
#> 2 4984859173592703 48Merge data
to join the data from different tables the recommended variable is
id, which is a unique 16 digit code.
artigos_publicados2 |>
dplyr::left_join(livros_publicados2) |>
dplyr::left_join(capitulos_livros2)
#> # A tibble: 2 × 4
#> id artigos livros capitulos
#> <chr> <int> <int> <int>
#> 1 3051627641386529 101 45 81
#> 2 4984859173592703 91 8 48Add information from a different tables.
artigos_publicados2 |>
dplyr::left_join(livros_publicados2) |>
dplyr::left_join(capitulos_livros2) |>
dplyr::left_join(dados_gerais |> dplyr::select(id, nome_completo)) |>
dplyr::select(nome_completo, artigos, livros, capitulos)
#> # A tibble: 2 × 4
#> nome_completo artigos livros capitulos
#> <chr> <int> <int> <int>
#> 1 Antonio Marcio Buainain 101 45 81
#> 2 Jose Maria Ferreira Jardim da Silveira 91 8 48Export to RIS format
writePublicationsRis(artigos_publicados,
filename = '~/Desktop/artigos_nome_citacao.ris',
citationName = T,
append = F,
tableLattes = 'ArtigosPublicados')
# full author name, ex: Antonio Marcio Buainain
writePublicationsRis(artigos_publicados,
filename = '~/Desktop/artigos_nome_completo.ris',
citationName = F,
append = F,
tableLattes = 'ArtigosPublicados')
writePublicationsRis(livros_publicados,
filename = '~/Desktop/livros.ris',
append = F,
citationName = T,
tableLattes = 'Livros')
writePublicationsRis(capitulos_livros,
filename = '~/Desktop/capitulos_livros.ris',
append = T,
citationName = F,
tableLattes = 'CapitulosLivros')